はじめに
ROSALINDの問題(Counting DNA Nucleotides)をやってみました。
問題は、1000 bp程度の塩基配列があってA, C, G, Tがそれぞれいくつあるかカウントして提出してください。というもの
ROSALINDがなにかわからない人や、まだ1問も解いていない人は下記記事を先に見てください。

Counting DNA Nucleotidesの回答方法はA, C, G, Tの順番でスペース区切り。順番でどの塩基か判断するので注意してください。私は順番を間違えてしまい、コレクトできず焦りました。
せっかくなのでバイオインフォマティシャンっぽく?biopythonを用いてカウントしてみた。
下記にコードを載せていますが、提出できるような形で公にしないほうが良いそうなので、C, Gのみカウントしています(A, Tを同じように入れればすぐ回答できますが……)。
#問題のデータセットを開く
with open("/home/bio/Downloads/rosalind_dna.txt", "r") as f :
dna_seq = f.read()
#ちゃんと開けるか確認
print(dna_seq)
#biopythonのSeqを使えるようにする
from Bio.Seq import Seq
#biopythonのSeqとして扱う
bp_dna_seq = Seq(dna_seq)
print(bp_dna_seq)
#.count()を使用してCをカウント
count_c = dna_seq.count("C")
print(count_c)
#同じように.count()を使用してGをカウント
count_g = dna_seq.count("G")
print(count_g)
#[]を使用してリストに入れる
count_list = [count_c, count_g]
#数値で.joinをしようとすると怒られるので map()を使用してstr (string)型に変更
map_list = map(str,count_list)
#.joinを使用する。区切り文字をスペースにして(ダブルクオーテーションの間を半角スペースする。","とかにすれば,で区切れる)結合
result = " ".join(map_list)
print(result)
#with openの"w"で"dna_count_result.txt"というファイルを出力する。中身は上にあるresultの内容
with open("dna_count_result.txt", "w") as f:
f.writelines(result)こんな感じでデータセットをダウンロードして解析し、出力されたファイルを提出すればクリアです。
回答がコレクトされると他の人の回答も見れるようになって、こんな汚いコードを書かなくても、もっとスマートなコード(回答)を見ることができます。
jupyter-labを使用した場合
ちなみにjupyter-labを使ってやると下記のようになり、こういった出力を確認しながら行う作業はやりやすいかもしれません。
実行前
実行後
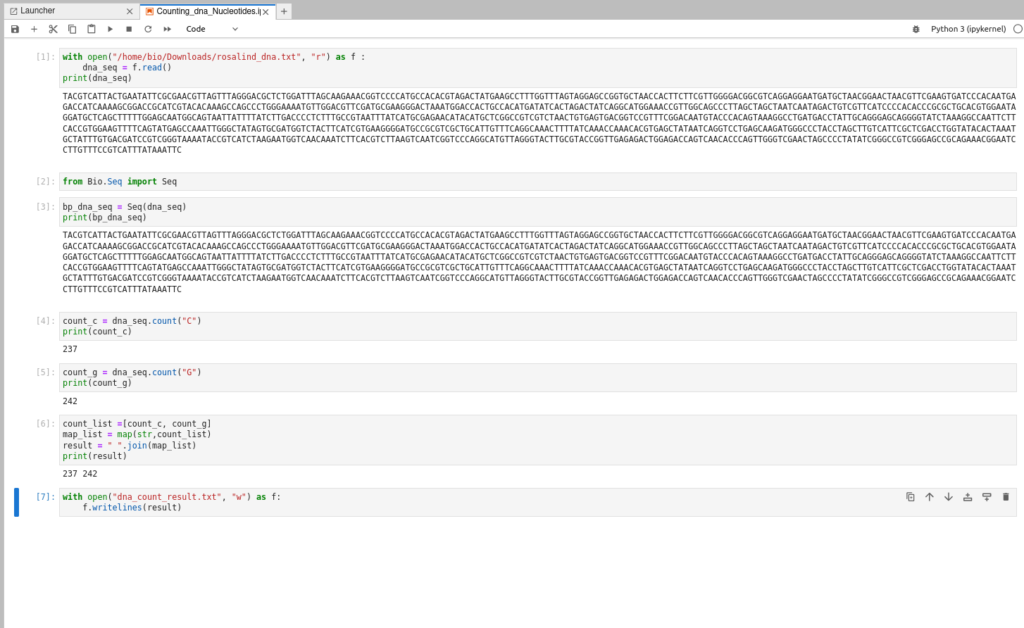
まとめ
こんな感じで問題を解いていきますが、実際に使用するとしたら欠損値、ギャップなども考慮しないといけないですね。
pythonやそもそもプログラミングをしたことがない人は、この記事を見てもよくわからないかもしれませんが、コマンドを一つひとつ調べながら実行していけばそのうちある程度はできるようになると思います。ウエットの実験ばっかりしてドライの実験をサボるとすぐに忘れていくので、バイオインフォマティクスは日々コツコツと勉強しないといけないですね。本当に……。


コメント