はじめに
Assembly(アセンブリとかアッセンブリ、アッセンブリーと呼んでる)にはレベルがあってややこしい。バイオインフォマティクスを独学で学んでいる人なら必ず混乱するところだと思う。
Assembly Levelを端的に表すなら下記のようになり、右に行くほど配列が長くなり、本来のゲノム情報に近づく
Read→Contig(コンティグ)→Scaffold(スキャフォールド)→Chromosome→Complete≒元の配列
これさえ頭に入っていれば普段の解析に大きな影響はないと思うが、ContigとScaffoldの違いが気になり、少し調べてみた。
ContigとScaffoldの違い
リードはNGSから読んだ断片ってことはわかるし、Contigがそれらをアセンブリしてつなげたものであることも分かる。ただContigとScaffoldの違いはなにかと言われればContigよりScaffoldのほうが長いくらいの理解であった。
色々な文献等見てみたが、信頼性が高そうなNCBIに載っていたものを和訳すると
Scaffold – 配列コンティグが隙間なく連結され、足場が形成されていますが、足場はすべて未配置または未局在です。
Contig – 配列コンティグのレベル以上には何も組み立てられていない。
となっていました。正直この説明だとよくわからないのですが、一言で表すなら
Contigをペアエンドのリード情報使用して繋げたものがScaffold
ってことだと思います。
ReadがCompleteになるまでの図
頭の中を整理するためReadがCompleteになるまでの図を作りました。
(これで正しいかわかりませんが……)
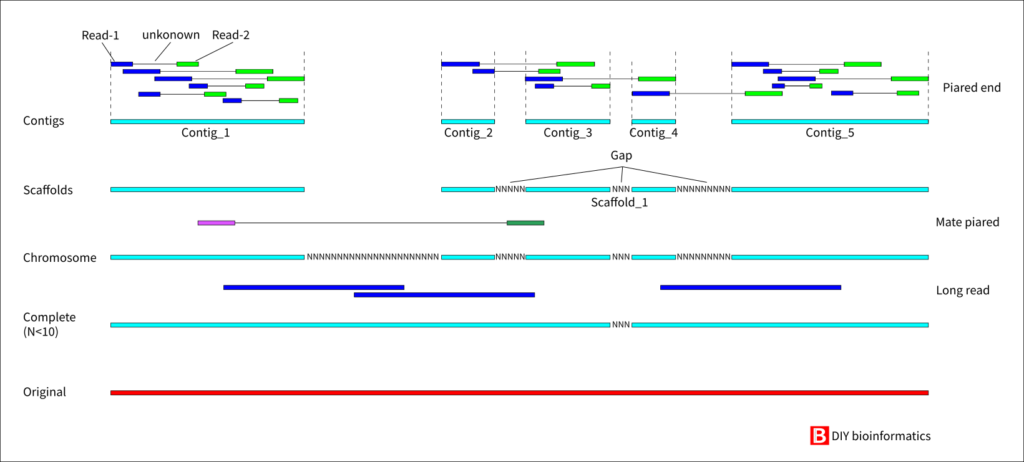
Readの情報からContigを作成し、次にPiared end(同じエリアから二種類の配列が取得でき、それらはキットや装置にもよるが1000 bp以下の範囲に存在する)であることを利用し、Contigsの位置関係を決め、間はNで埋めてScaffoldとする(Nの個数はソフトウェア上で適当な数にする?)。Chromosomeレベルにしたい場合、ほとんどのケースで、繰り返し配列やGC含量などの関係からMate piaredかLong readまたはその両方を使用して一本にする必要がある。その過程でNが9以下になりCompleteになることもあるし、カバレッジやGapを埋めるため、再度Piared endのシーケンスを行い、Completeの配列にしたりする。
アセンブルしたContigとScaffoldの結果を詳細に見てみると、Nを除く前は塩基数はScaffoldのほうが多く、Nを除くとContigのほうが塩基数が多くなる。おそらくペアエンドの情報を使うことで、カバレッジが得られてGapにならず、つながる箇所があるのかもしれない。ほぼ誤差ですが。
Assembly Levelにおける注意点
調べていて、注意しなくてはいけないと再認識したこと
- CompleteでもNが存在する可能性があること
- ショートリードでは繰り返し配列をうまく読めないこと
- 同じPiared endからアセンブリしたら、ContigとScaffoldの差は基本的に位置関係の情報のみ
まとめ
要するに細かいこと気にしなくていい解析であれば
read→contig→scaffold→chromosome→complete≒元の配列
ってこと。


コメント